几种交叉验证 cross_validation
- 计算机科学
- 2023-04-21
- 199热度
- 0评论
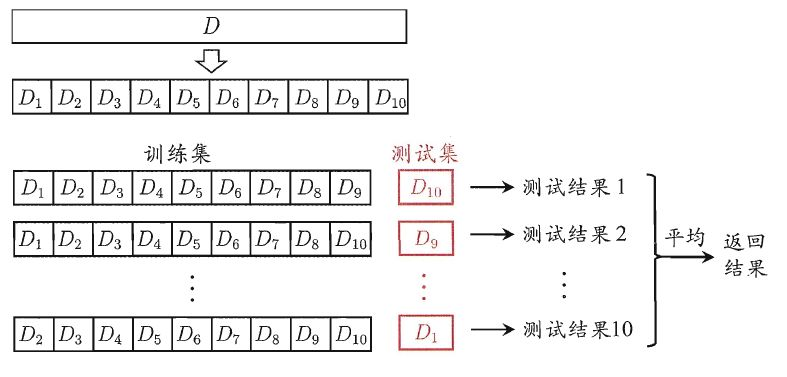
1、交叉验证概念:
将一个初始数据集生成多个训练-测试切片,使用切片去调整模型
2、The Validation Set Approach(train-test split)
数据集随机划分训练集和验证集,最常使用,直接划分数据集方法
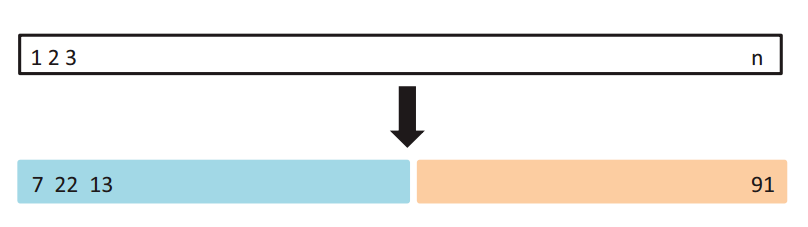
缺点:不均匀数据集不适合;一大块没有训练机会
优点:简单易执行
from sklearn.model_selection import train_test_split
# df数据集,X是特征,y是标签
X=df.data
y=df.target
# 直接划分成训练集的特征与标签,测试集的特征与标签
X_train,X_test,y_train,y_test=train_test_split(X,y,test_size=0.2,random_state=123)2、K折交叉验证 K-Fold Cross-Validation
数据集划分为K个大小相同的部分,用一个作为验证,K-1个作为训练,最终准确度是通过取k个平均值来确定的
缺点:不均匀数据集不适用;不适用于时间序列
from sklearn.model_selection import cross_val_score
from sklearn.model_selection import KFold
X = pd.data
y = pd.target
kf = KFold(n_splits=5)
# knn是用来验证的模型
score = corss_val_score(knn,X,y,cv=kf)3、分层K折交叉验证(Stratified K_Fold Cross-Validation
K折的升级版,每一分层里面又进行了分层
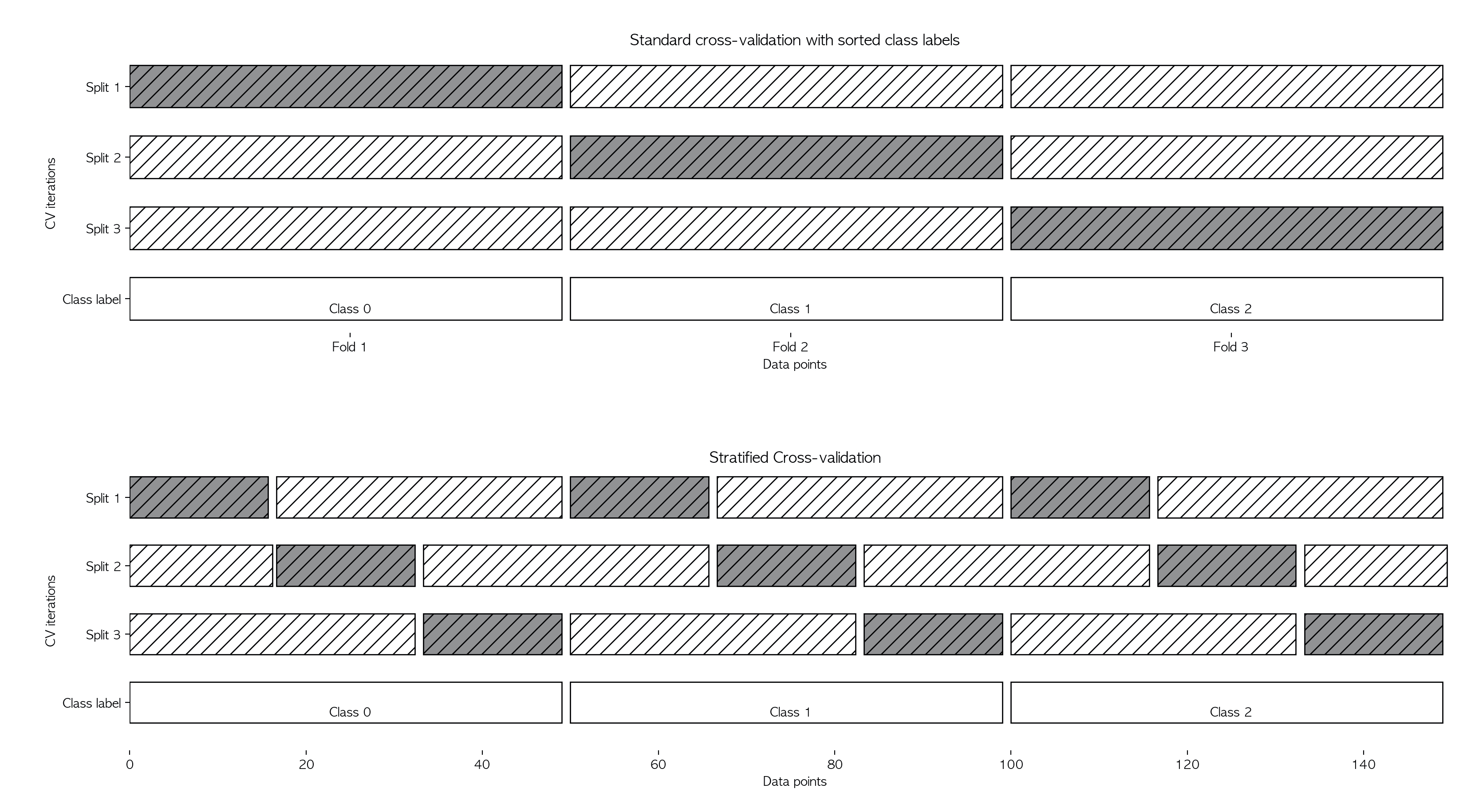
缺点:不适合时间序列
优点:适用于不均匀数据集
from sklearn.model_selection import cross_Val_score,StratifiedKFold
X = df.data
y = df.target
stratifiedkf=StratifiedKFold(n_splits=5)
score = cross_val_score(model,X,y,cv=stratifiedkf)4.1、Leave One Out cross-validation
留下一个验证,其余用来训练
from sklearn.model_selection import LeaveOneOut,cross_val_score
X = df.data
y = df.target
loo = LeaveOneOut()
score = Cross_val_scoer(model,X,y,cv=loo)4.2、Leave P out cross-validation
留下P个样本,n-p个进行验证
缺点:速度慢、不适合不均匀数据集
优点:用上了所有的数据
from sklearn.model_selection import LeavePOut,cross_val_score
X=df.data
y=df.target
lpo=LeavePOut(p=2)
lpo.get_n_splits(X)
# 注意,这里的tree是验证的模型对象
score=cross_val_score(model,X,y,cv=lpo)5、Monte Carlo Cross-Validation MCCV蒙特卡洛交叉验证
自由选择训练和验证集大小,样本可能会多次出现
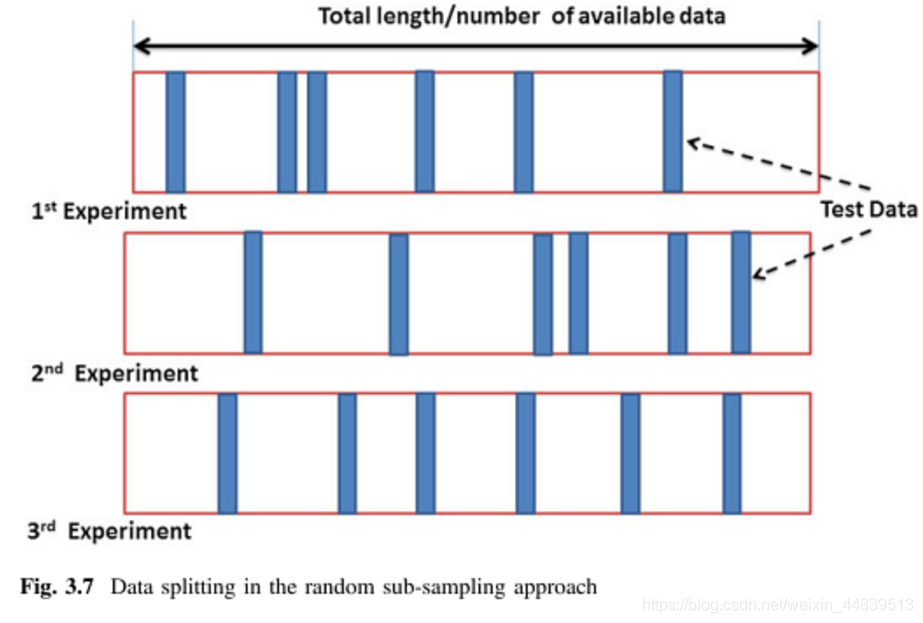
缺点:少数样本不会被训练、不适合不平衡数据集
优点:不再依赖重复的fold数量
from sklearn.model_selection import shuffleSplit,cross_val_score
shuffle_split = ShuffleSplit(test_size=0.3,train_size=0.5,n_split=10)
score = cross_val_score(model,X,y,cv=shuffle_split)6、Time Series Cross-Validation 时间序列交叉验证
从小的子集开始,接着对后续的样本进行预测
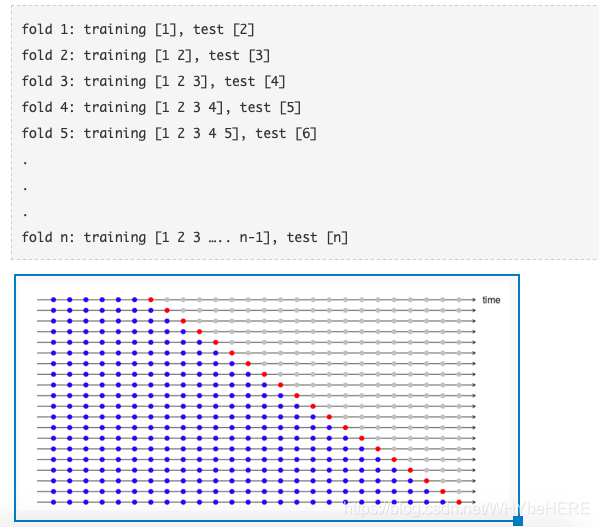
from sklearn.model_selection import TimeSeriesSplit
X = np.data
y = np.traget
time_series = TimeSeriesSplit()
print(time_series)
for train_index, test_index in time_series.split(X):
print("TRAIN:", train_index, "TEST:", test_index)
X_train, X_test = X[train_index], X[test_index]
y_train, y_test = y[train_index], y[test_index]一些Q & A
https://zhuanlan.zhihu.com/p/128984157
https://zhuanlan.zhihu.com/p/24825503
- 为什么有的人说随机森林不需要用交叉验证呢?
因为随机森林在训练时会存在袋外误差的计算,这个误差的计算和交叉验证非常相似,所以有人会说随机森林不需要交叉验证
- 请问,比如十折交叉检验,每次随机划分9份训练,一份测试,这个过程做10遍。这样基于不同的训练集,实际最后应该得到了10个不同的模型。然后最终模型是选择10次测试中结果最优的那个模型,对么?如果是,那么求解10次测试结果平均值的意义在哪里?这样做是为了评估做交叉检验之前已经建好的那个模型么?另外,是否可以理解为,交叉检验本身就涵盖了训练建模的过程了呢?
我的理解是,你这十遍都是用的同一套超参数,是用来评估该套超参数(不是模型权重)的性能(根据泛化误差的大小),每遍权重都是随机初始化的,最终模型这十个那个都不选,将所有数据重新训练(可以在原先权重基础上),得到的模型才是最好的模型
- 在scikit-learn的框架上写的是把数据集分成测试集和训练集,测试集是永远不能动的,把训练集分成训练子集和测试子集,用K-fold评估并且选择最优的模型以及相应参数,最终在测试集上进一步测试。可参考scikit-learn.org/stable
交叉验证的这个手段一般可用于模型的“选择”,通过交叉验证后学习器的综合表现来选择不同的模型和超参数
- K-fold划分数据进行训练有k个训练模型,那最终选取哪个模型?还有为什么要计算所有模型的平均误差?
这里有个误区,我们进行k-fold是将数据划分为k折,每次取k-1的数据训练然后在剩下的一份验证打分,这样经过k轮后就得到一个模型的k个得分(注意“模型”只有一个,当你确定具体算法和参数组合以后就确定了模型,而模型拟合数据得到的是学习器(比如分类器,你可以理解为模型的具体实例)),那为什么要取平均呢,其实在前面说过单独取部分数据作为验证集来验证可能结果不稳定,不具代表性,所以我们做了k次验证,而且每次以k-1组数据训练、剩余一组验证就保证我们把原始的训练集都使用了一遍,每个数据都作为训练数据、测试数据被使用过,现在对这k个学习器的得分取个平均分来代表这个模型的表现就有说服力了,而对于其他模型(不同模型比如SVM和决策树或者统一模型的不同参数组合)分别进行k折交叉验证,每一个模型都得到各自学习器的平均得分,我们通过比较这些模型的平均得分就能知道最佳的一个模型是哪一个,最终就把这个模型(算法及参数组合)拿出来重新用全部训练集训练得到最后的学习器,然后对测试数据作出预测,整个过程大概是这样,注意区分“模型”与“学习器”,“参数”与“超参数”
是对的,你只要知道这里调的参数不是指算法中的“参数”比如线性模型的权重,而是一些模型特有的超参数,比如SVM的核函数选择、集成模型比如随机森林的最小分割子树之类的
可以这么理解,举经典的考试的例子,测试集相当于期末考试,试卷的题目你是无法预先知道的,但你想对自己的能力做一个自我检测,于是设置了验证集(交叉验证相当于充分利用训练集分组进行多次验证);所以交叉验证只是你自己对自己的模型有一个评估,在评估中可以查缺补漏(调参、选最佳学习器),最后你拿最佳的参数组合用所有的训练集拟合出你的终极学习器,去参加考试(在测试集上作出预测);从这个角度来看交叉验证的平均准确率本身与测试机的准确率是无关的,我们希望通过手头拥有的数据来模拟出一个自我的评价,也就是希望这个交叉验证的结果能够代表模型在真实测试集的表现
#求a最大值的位置
np.where(a = np.max(a))
#生成1-11
a = np.arange(1,11)
#数组数量
a.size
#最大值
a.max()
#图像清晰度
plt.figure(dpi=100)
#解决中文不显示的问题
matplotlib.rcParams['font.sans-serif'] = ['SimHei'] # 显示中文
# 为了坐标轴负号正常显示。matplotlib默认不支持中文,设置中文字体后,负号会显示异常。需要手动将坐标轴负号设为False才能正常显示负号。
matplotlib.rcParams['axes.unicode_minus'] = False
